如题,我在用scrublet进行单细胞预测的时候,按照默认的参数去跑,得到了umap的预测图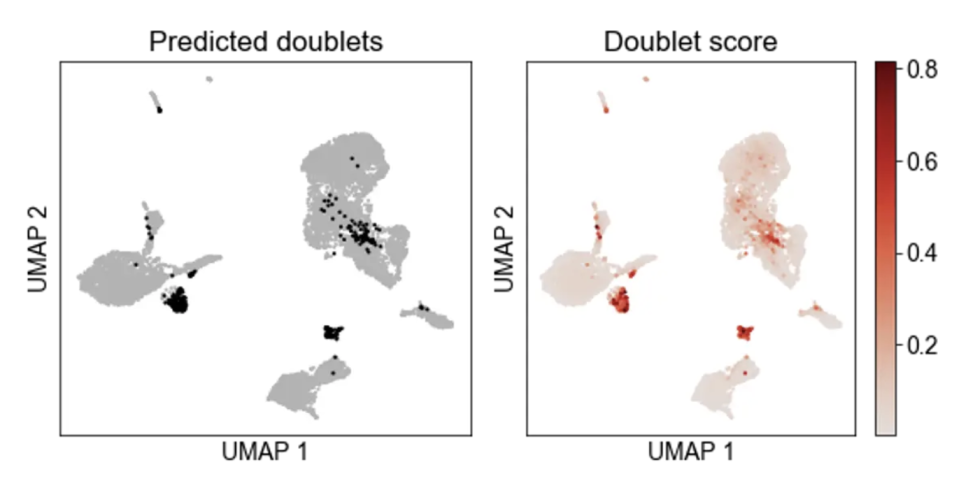
教程里说黑色的点就是预测的双细胞,但是当我使用以下代码导出文本准备导入seurat去筛选的时候,我发现在这个doublet.txt的文件中,predicted_doublets这一列,所有细胞都是False,按理说在上图中所出现的细胞就应该是TRUE,请问这是怎么回事呢??
out_df['doublet_scores'] = doublet_scores
out_df['predicted_doublets'] = predicted_doublets
out_df.to_csv(input_dir + '/doublet.txt', index=False,header=True)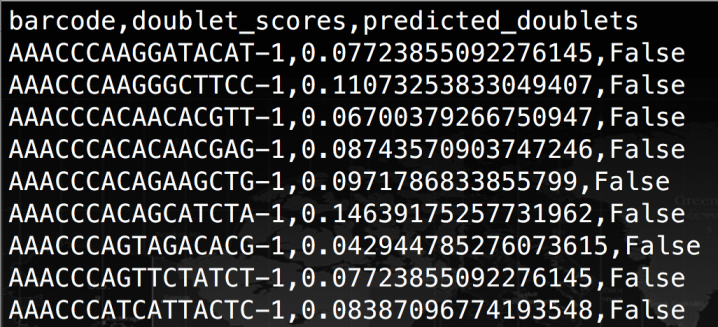
1 回答
找一个可以记录问题的地方