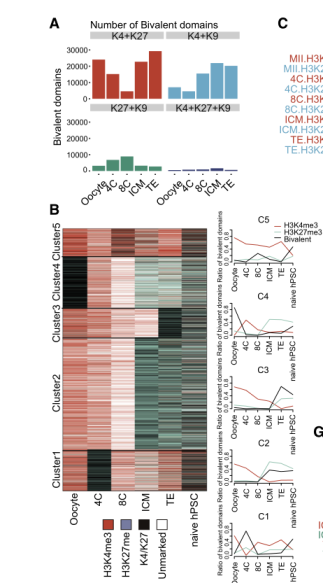
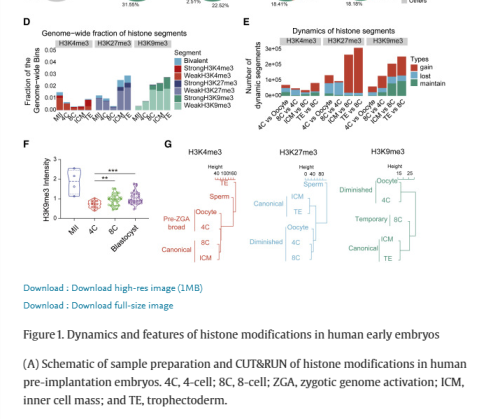
Bivalent domains是指同时存在H3K4me3和H3K27me3修饰的染色质区域,它们在基因调控中发挥重要的作用。下面简单介绍一些常用的计算方法。
一、ChromHMM
ChromHMM是一种常用的计算bivalent domains的工具。其基本思路是将染色质区域划分为不同的状态(如active promoter, enhancer, bivalent promoter等),再根据H3K4me3和H3K27me3的分布情况确定bivalent domains的位置。具体的计算流程可以参考ChromHMM官方文档。
二、MACS2
MACS2是一种常用的ChIP-seq分析工具,它可以用来计算H3K4me3和H3K27me3的峰值。根据峰值的分布情况,可以确定bivalent domains的位置。具体的计算流程可以参考MACS2官方文档。
三、HOMER
HOMER是一种常用的染色质分析工具集,其中包含了多种计算bivalent domains的工具。比如,可以使用annotatePeaks.pl命令将H3K4me3和H3K27me3的信号合并起来,然后根据信号的分布情况确定bivalent domains的位置。具体的计算流程可以参考HOMER官方文档。
以上是一些常用的计算bivalent domains的方法,具体的选择可以根据实验设计和数据分析的需求来确定。
阅读全文
收起全文