
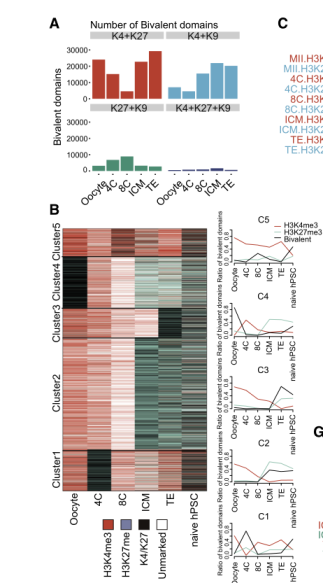

还有下面这种不同状态的分析,感觉像ChromHMM 分析的又好像不是。
图片是来自同济大学高绍荣团队发的文章,https://www.sciencedirect.com/science/article/pii/S1934590922002508?via%3Dihub
试着发邮件要了下画图数据分析过程和代码也没有回复
文章说计算来自一篇cell 文章https://www.sciencedirect.com/science/article/pii/S0092867406003801?via%3Dihub

挺想做上面这些分析的,哪位大佬能搞个详细教程呀。
这个所谓的bivalent domain指的就是一段染色质区域,处于即可能开放又可能抑制的状态。
比如你上面提到的,H3K4me3这个就是典型的active的 histone marker;
H3K27me3就是repressive的 histone marker;
然后如果这两个同时存在,就可以定义为bivalent domain。
看你上面的截图,就应该是先把基因组分区域,分区域的方法通常是分bin,然后你就可以计算这一个bin区间里的H3K4me3和H3K27me3的信号强度,两个都存在的时候就可以定义。
我看你上面还提到了chromHMM,用这个也可以,就是用chromHMM分出来H3K4me3和H3K27me3同时存在的那个类型就定义为bivalent domain即可。这个和直接分bin自己算coverage类似,没太大差别。
这家伙很懒,还没有设置简介