在WGCNA分析中,纳入了两种疾病的两套数据,构建了一致性(concensus)网络识别模块儿。
得出至少跟一种疾病相关的模块儿之后(pearson correlation p<0.05),如何在其中寻找hub基因?
传统的分析可以根据GS(基因和性状相关性)和ME(基因和模块儿相关性)等指标来筛选,我的网络中存在多套数据,可以计算GS和ME的metaZ和metaP值,是否可以初步筛选GS和ME的metaP都小于0.05的基因呢?
-------------------------------------------------------------------------------------------------------------------
2023.05.18,追加:
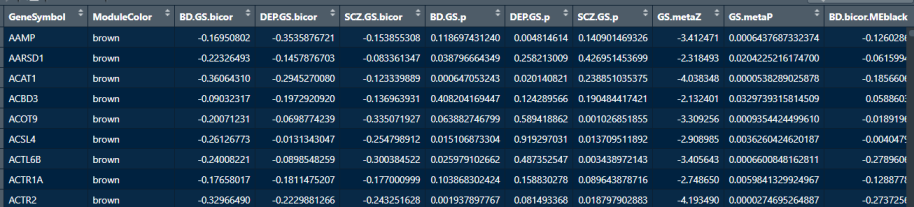
一下是我挑出来的brown module相关的结果,DEP, SCZ, BD为三种疾病表型,数据中包括模块儿中的基因,基因与某表型的相关结果(如BD.GS.cor/p),某表型基因表达谱和模块的相关结果(如BD.cor/p.MEred),以及两种结果的metap/Z。
在WGCNA分析中,一致性网络构建完成后,我们可以使用多种方法来筛选hub基因。以下是一些可能的方法:
综上所述,我们可以选择以上任意一种或多种方法来筛选WGCNA分析得出的consensus module中的hub基因。在选择方法时,需要考虑数据的特点和研究目的,并进行合理的比较和分析。
99年bioinfo嘿嘿嘿