问题:
在做WGCNA时,已经筛选到感兴趣的模块,假设是“yellow”,而且这个模块我已经得到他的TOM矩阵,是68x68的矩阵,下面只展示了6行6列的数据。
modTOM[1:6,1:6]
Mgp Vtn Atp13a5 Abcc9 Slc6a20a Lin7a
Mgp 0.00000000 0.08310508 0.05093996 0.04937136 0.05717230 0.01967644
Vtn 0.08310508 0.00000000 0.14591322 0.13633820 0.15664908 0.08961941
Atp13a5 0.05093996 0.14591322 0.00000000 0.09803593 0.11624331 0.06986203
Abcc9 0.04937136 0.13633820 0.09803593 0.00000000 0.10219968 0.06263205
Slc6a20a 0.05717230 0.15664908 0.11624331 0.10219968 0.00000000 0.07074445
Lin7a 0.01967644 0.08961941 0.06986203 0.06263205 0.07074445 0.00000000
然后我使用如下代码获得两个可以在cytoscape中使用的文件:
cyt = exportNetworkToCytoscape(
modTOM,
edgeFile = paste("CytoscapeInput-edges-", paste(module, collapse="-"), ".txt", sep=""),
nodeFile = paste("CytoscapeInput-nodes-", paste(module, collapse="-"), ".txt", sep=""),
weighted = TRUE,
threshold = 0.005,
nodeNames = modProbes,
nodeAttr = modulecolors[inModule]
)
接下来我下载了cytoscape,并且导入了如下的文件(通过上面代码得到的文件)
然后我在cytoscape上,将上面的文件设置了源节点和靶节点,最终我得到的结果图是下面这样的
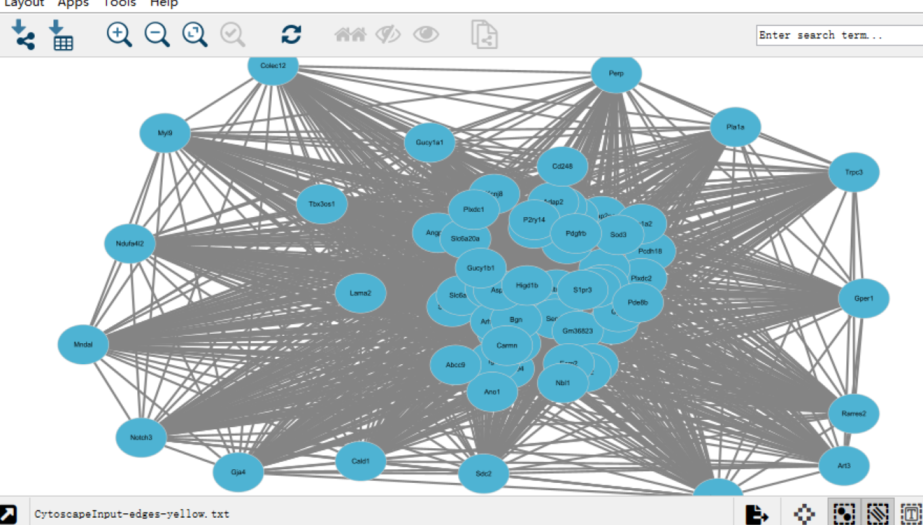
然后后面我就不知道该如何寻找hub基因了?
或者各位大神有其他方法找hub基因的话,也请赐教,感谢!
1 回答